Cloud computing and digital transformation have been powerful enablers for genomics. Genomics is expected to be an exabase-scale big data domain by 2025, posing data acquisition and storage challenges on par with other major generators of big data. Embracing digital transformation offers a practically limitless ability to meet the genomic science demands in both research and medical institutions. The emergence of cloud-based computing platforms such as Microsoft Azure has paved the path for online, scalable, cost-effective, secure, and shareable big data persistence and analysis with a growing number of researchers and laboratories hosting (publicly and privately) their genomic big data on cloud-based services.
At Microsoft, we recognize the challenges faced by the genomics community and are striving to build an ecosystem (backed by OSS and Microsoft products and services) that can facilitate genomics work for all. We’ve focused our efforts on three main core areas—research and discovery in genomic data, building out a platform to enable rapid automation and analysis at scale, and optimized and secure pipelines at a clinical level. One of the core Azure services that has enabled us to leverage high performance compute environment to perform genomic analysis is Azure CycleCloud.
Galaxy and Azure CycleCloud
Galaxy is a scientific workflow, data integration, and data analysis persistence and publishing platform that aims to make computational biology accessible to research scientists that do not have computer programming or systems administration experience. Although it was initially developed for genomic research, it is largely domain agnostic and is now used as a general bioinformatics workflow management system. Galaxy system is used for accessible, reproducible, and transparent computational research.
- Accessible: Programming experience is not required to easily upload data, run complex tools and workflows, and visualize results.
- Reproducible: Galaxy captures information so that you don’t have to; any user can repeat and understand a complete computational analysis, from tool parameters to the dependency tree.
- Transparent: Users share and publish their histories, workflows, and visualizations via the web.
- Community-centered: Inclusive and diverse users (developers, educators, researchers, clinicians, and more) are empowered to share their findings.
Azure CycleCloud is an enterprise-friendly tool for orchestrating and managing high-performance computing (HPC) environments on Azure. With Azure CycleCloud, users can provision infrastructure for HPC systems, deploy familiar HPC schedulers, and automatically scale the infrastructure to run jobs efficiently at any scale. Through Azure CycleCloud, users can create different types of file systems and mount them to the compute cluster nodes to support HPC workloads. With dynamic scaling of clusters, the business can get the resources it needs at the right time and the right price. Azure CycleCloud automated configuration enables IT to focus on providing service to the business users.
Deploying Galaxy on Azure using Azure CycleCloud
Galaxy is used by most academic institutions that conduct genomic research. Most institutions that already use Galaxy want to stick to it because it provides multiple tools for genomic analysis as a SaaS platform. Users can also deploy custom tools onto Galaxy.
Galaxy users generally use the SaaS version of Galaxy as part of UseGalaxy resources. UseGalaxy servers implement a common core set of tools and reference genomes and are open to anyone to use. All information on its usage is available on the Galaxy Platform Directory.
However, there are some research institutions that intend to deploy Galaxy in-house as an on-premises solution or a cloud-based solution. The remainder of this article describes how to deploy and run Galaxy on Microsoft Azure using Azure CycleCloud and grid engine cluster. The solution was built during the Microsoft hackathon (October 12 to 14, 2021) with code implementation assistance from Azure HPC Specialist, Jerry Morey. The architectural pattern described below can help organizations to deploy Galaxy in an Azure environment using CycleCloud and a scheduler of choice.
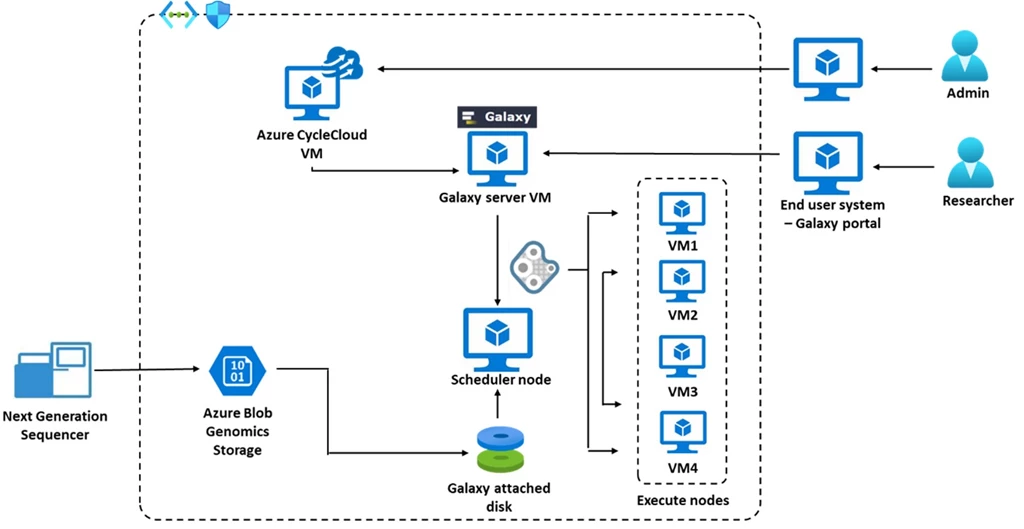
As a pre-requisite, genomic data should be available in a storage location, either cloud or on-premises. Azure CycleCloud should be deployed using the steps described in the “Install CycleCloud using the Marketplace image” documentation.
Cluster deployment that is truly supported by Galaxy on the cloud is called the unified method. In this method, the copy of Galaxy on the application server is the same copy as the one on the cluster nodes. The most common method to do this would be to put Galaxy in a network file system (NFS) somewhere that is accessible by the application server and the cluster nodes. This is the most common deployment method for Galaxy.
An admin user can SSH into Azure CycleCloud virtual machines or Galaxy server virtual machines to perform admin-related activities. It is recommended to close the SSH port when in production. Once the Galaxy server is running on a node, end users (researchers) can load the portal on their end device to perform analysis tasks which include loading data, installing, uploading tools, and more.
Access to functionalities (such as installing and deleting tools versus the usage of tools for analysis) are controlled by parameters defined in galaxy.yml that resides in the Galaxy server. Once a user accesses a functionality, they are converted to jobs that are submitted to the grid engine cluster for further execution.
Deployment scripts are available to ease deployment. These scripts can be used to deploy the latest version of Galaxy on Azure CycleCloud.
Following are the steps to use the deployment scripts:
- Git clone this project (The project is in active development, so cloning the latest release is recommended).
git clone –b release_21.09 https://github.com/themorey/galaxy-gridengine.git
- Upload project to CC locker.
cd galaxy-gridengine
Modify files (if needed)
cyclecloud locker list
Azure cycle Locker (az://mystorageaccount/cyclecloud
cyclecloud project upload "Azure cycle Locker"
- Import cluster template to CC.
cyclecloud import_cluster -c -f templates/gridengine-galaxy2.txt
NOTE: Substitute with a name for your cluster—all lower case, no spaces.
- Navigate to CC Portal to configure and start the cluster.
Wait for 30 to 45 minutes for the Galaxy server to be installed.
To check if the server is installed correctly, SSH into Galaxy server node and check galaxy.log in /shared/home/ directory.
This deployment was adopted by a leading United States-based academic medical center. The Microsoft Industry Solutions team helped deploy this solution on the customer’s Azure tenant. Researchers at the center tested to assess the parity of this solution to existing Galaxy deployment on their on-premises HPC environment. They were able to successfully test the deployed Galaxy server that used Azure CycleCloud for job orchestration. Several common bioinformatics tools such as bedtools, fastqc, bcftools, picard, and snpeff were installed and tested. Galaxy supports local user by default. As part of this engagement, a solution to integrate their corporate active directory was tested and deployed. The solution was found to be on par with their on-premises deployment. With the increased number of execute nodes and size of those nodes, they found that the jobs were executed in less time.
For more information, support, or guidance related to the content in this blog, we recommend you reach out to your Microsoft sales representative.
Learn more
Learn more about Microsoft Genomics solutions.
- Microsoft Genomics service on Azure.
- Azure CycleCloud—HPC Cluster and Workload Management.
- Galaxy on Azure deployment scripts.
